新闻中心
NEWS
【文章分享】 重症监护脓毒症患者阳性血培养的快速纳米孔测序和预测性药敏试验

中文标题:重症监护脓毒症患者阳性血培养的快速纳米孔测序和预测性药敏试验
杂志全称:《Microbiology Spectrum》
影响因子:9.043
发布时间:2024年01月09日
原文链接:https://doi.org/10.1128/spectrum.03065-23
研究背景 >>
脓毒症是导致疾病和死亡的主要原因,快速的病原体鉴定和微生物药敏性表型分析对于选择治疗方案和确保病患安全至关重要。目前的病原体鉴定和基于培养的药敏试验(Antimicrobial Susceptibility testing,AST)可能需要三天或更长时间,在本研究中,研究者旨在确定牛津纳米孔(Oxford Nanopore Technologies,ONT)测序是否可以在保证诊断性能的前提下减少临床报告的生成周期。
研究方法 >>
整个实验流程如图1所示,实验选取的研究对象是以脓毒症为临床特征的已入住重症监护室的儿童和成人。首先是去除宿主基因组DNA;然后进一步提取微生物gDNA,随后是DNA质量和纯度检查。使用ONT基因组DNA连接试剂盒制备文库,并在MinION MK1B设备上运行,测序过程使用MinKNOW检测。文库运行72小时,但大多数样本在6-12小时内产生了足够的数据以供进一步分析,使用PycoQC进行QC分析。此外,从阳性血培(BC)菌液中分离的纯菌落也进行了全基因组测序,整个测序过程在Illumina MiniSeq平台上进行。
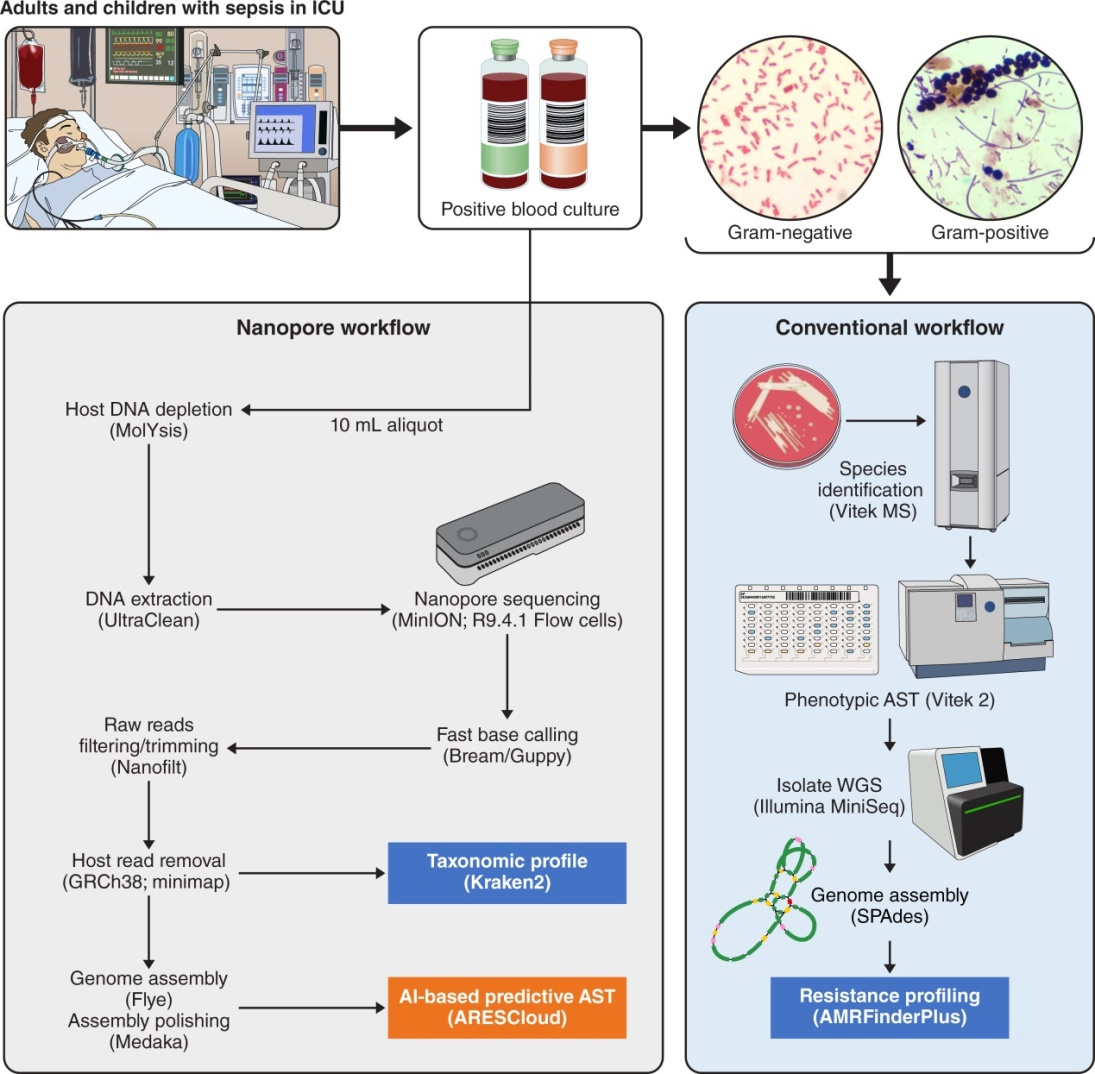
图1:Nanopore测序和阳性BC常规分析工作流程
数据分析和处理 >>
原始下机数据经过Nanofilt进行质量过滤,使用Minimap2对其进行了与参考基因组GRCh38的比对,以去除宿主基因组reads,使用Kraken2进行了分类学分配。同时将过滤下来的数据使用flye v2.9进行组装,使用参数“—nano-raw –meta –iterations 3”。使用MaxBin v2.2.7和MetaBAT v2.15来执行宏基因组分箱操作,然后使用DAS Tool v1.1.5来将不同宏基因组分箱后得到的bins进行整合,以提高对高质量未分箱contigs中特定抗微生物耐药性(Anti-microbial Resistance, AMR)基因的保留率。基因组组装完成后使用Checkm2来进行后续评估,组装结果上传到AREScloud进行药物敏感性预测。最后使用基于ARESdb训练的堆叠分类机器学习来预测AST模型,同时结合抗性预测的规则,通过ResFinder4执行物种特异性的敏感性和耐药性预测。作为参考,研究者还将来自BC的ONT测序耐药基因分型与通过Illumina测序的纯培养分离物进行比较,来确定体外耐药基因的基因型,并比较样本类型中AMR基因的存在/缺失情况的一致性。
实验结果 >>
血培养微生物鉴定
整个实验一共使用了来自201名患者的66份阳性血液样本,排除非细菌生长、缺失样本和外源污染物后,剩下52份用于测序分析,鉴定后分为27种革兰氏阳性菌和23种革兰氏阴性菌样本,以及2份多菌样本(表1)。

表1:常规血液培养后的物种鉴定
测序样本分类学鉴定和体外AST预测
排除污染物、厌氧生物以及存在多菌的样本之后,剩下42个样本进行了AST预测和AST表型比较。针对其中具有高质量AREScloud预测结果的24个样本,进行了262次AST预测,总体分类一致性(Categorical Agreement,CA)为89.3%,但极重大误差率(Very Major Error, VME)为12.1%,重大误差率(Major Error, ME)为10.5%(表2)。对于总体42个样本的所有470个AST预测,总体CA为87.7%,VME为28.4%,ME为8.3%(表3)。通过比较常规培养与测序分析两个方法,发现两个样本在属水平上一致,但测序在分类学鉴定上更为准确。
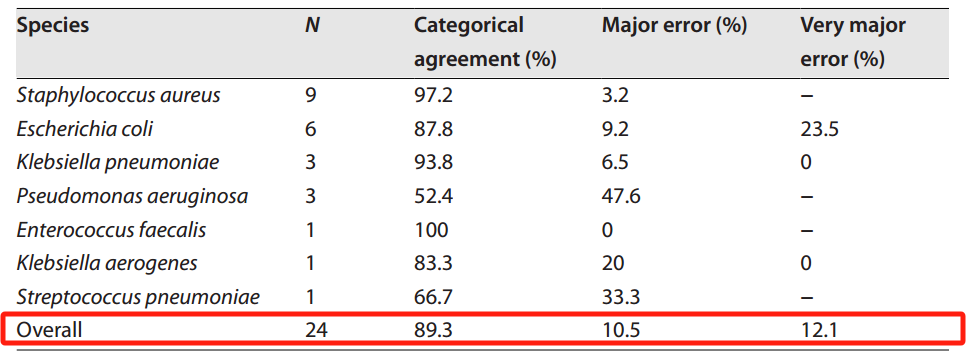
表2:ONT与常规培养illumina测序的预测AST表现(高质量样本)
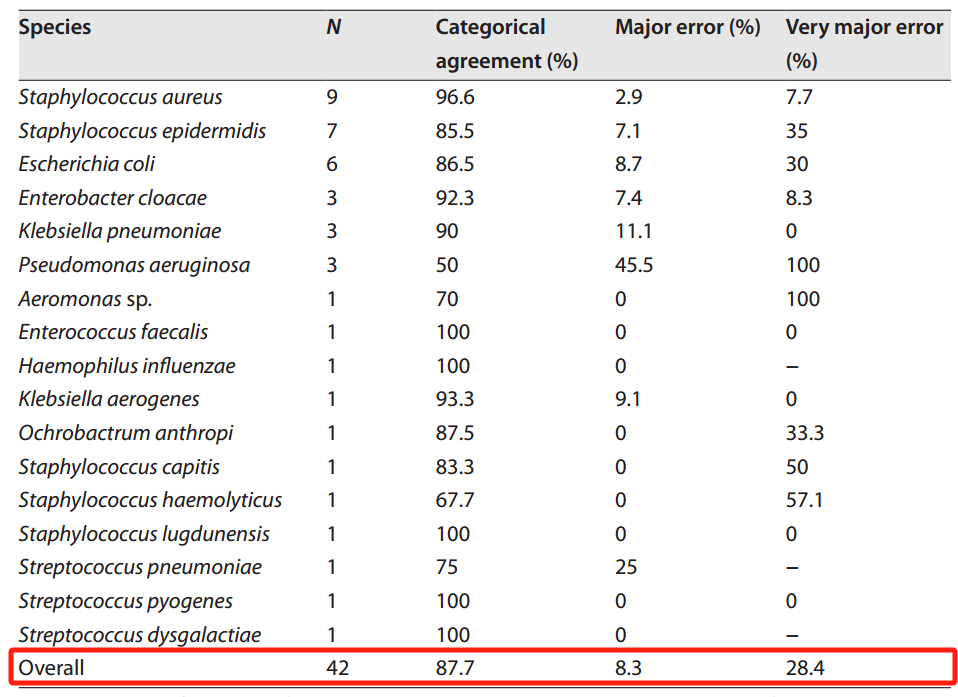
表3:ONT与常规培养基illumina测序的预测AST表现(所有样本)
ONT和illumina测序结果分析
比较基于血培养分离的ONT测序结果和基于纯培养分离的illumina测序结果的热图(见图2),发现两者AMR基因上出现了一些差异。基于illumina的纯分离物测序检测到了额外的AMR基因靶点。在单细菌的样本中,ONT技术检测到的AMR基因靶点比illumina多一个。
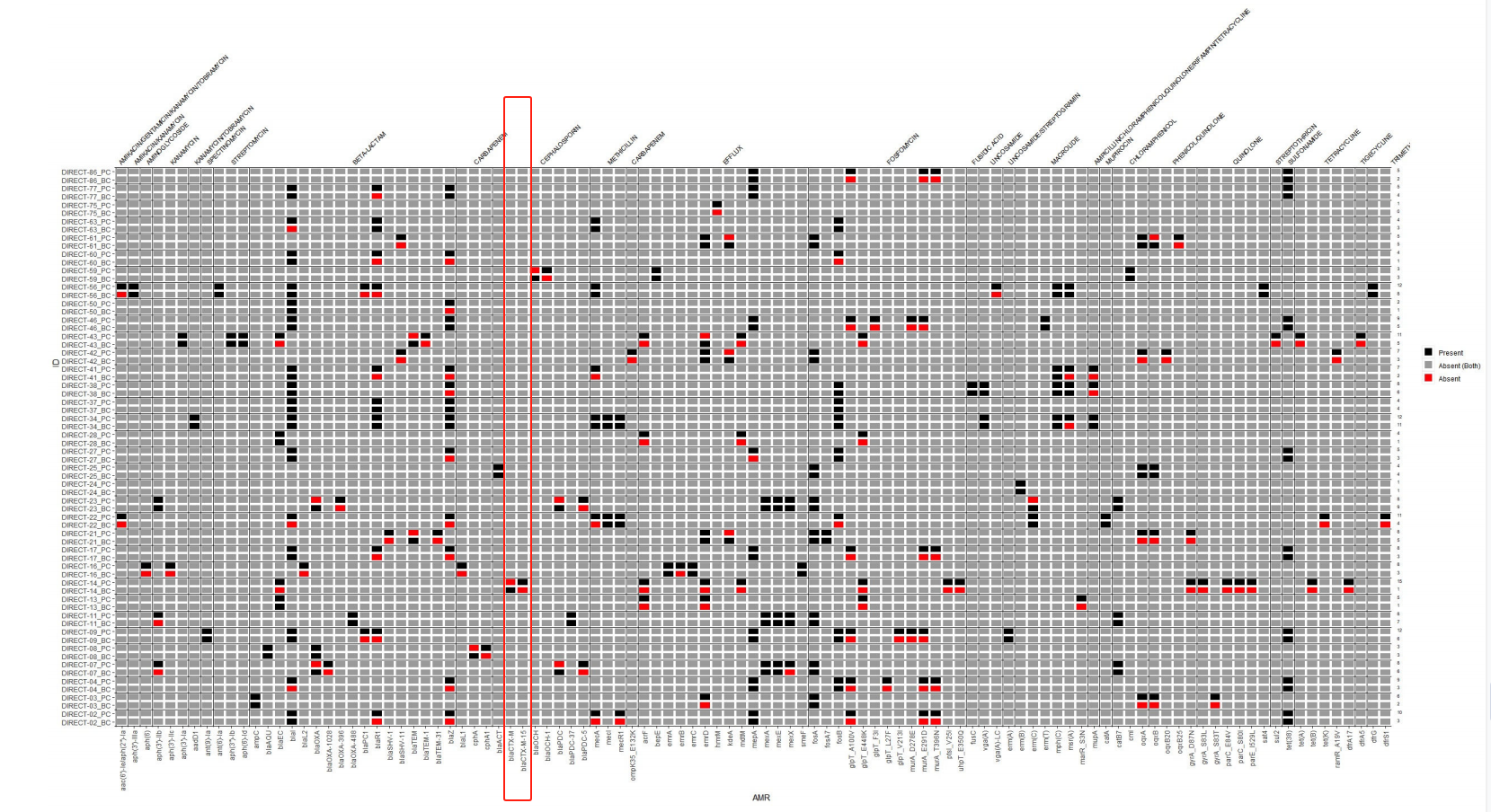
图2:ONT测序和illumina测序AMR基因热图比较
纵轴样本后缀“PC”表示illumina测序,“BC”表示ONT测序
报告周期
统计基于BC进行ONT测序的整个报告周期时间来看,测序前处理步骤需约4小时;对于单个样本,芯片运行约12小时;AREScloud的AST测试过程约1小时,整个路程在9-17小时内实现,比常规AST测试方法和基于 illumina的方法(长达48小时或更长)更快。
结论 >>
目前临床应用上有几种快速分子诊断试剂可用于物种鉴定和耐药基因检测(例如,BCID2,eplex,VERIGENE,T2MR等),但这些试剂通常限制了相对有限数量的物种和目的耐药基因。基于测序的方法的一个关键优势不仅在于可以检测样本中潜在的任何细菌,还能提供多种抗生素的预测表型,方便选择适当的治疗方案,可以最大限度的提高临床效用。
本研究证实阳性BC液直接测序是可行的,可以提供准确的病原微生物物种水平鉴定,基于ONT测序的方法可能更快,但由于本研究中常见菌群出现MEs/VMEs比例较大的情况,在考虑临床应用之前仍有很长一段距离。

电话:021-61283010
网址:www.bio-chain.com
邮箱:marketing@bio-chain.com
地址:上海市徐汇区虹漕路421号65幢303室
上海柏辰生物科技有限公司 版权所有 严禁复制 网站备案号:沪ICP备09099502号-1 沪公网安备 31010402002899号 网站建设:铭心科技



