新闻中心
NEWS
生信篇 | FASTA序列分析工具
生信专题 FASTA序列分析工具 ” 在对测序数据进行分析时,经常会碰到fasta文件的处理,如基因组组装、RNA-seq和蛋白质组学的分析等。使用现有的分析工具如SeqKit统计长度,往往需要手动筛选序列,再利用R或Python进行分析数据的可视化。这无疑是会导致操作的冗余且容易出错。本文介绍一款处理fasta数据的分析软件——SeqLengthPlot,其可将序列长度分析全流程(统计→筛选→可视化)整合为单一命令行工具,显著提升效率。
1

简介
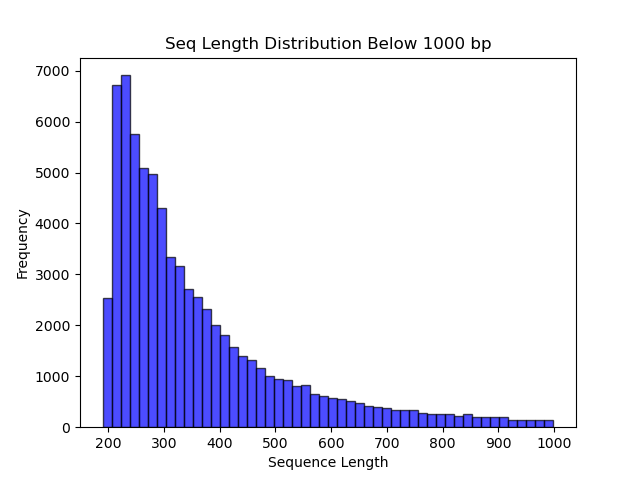
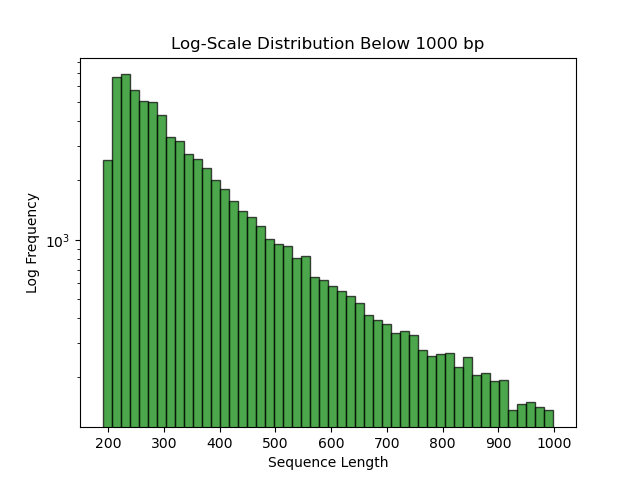
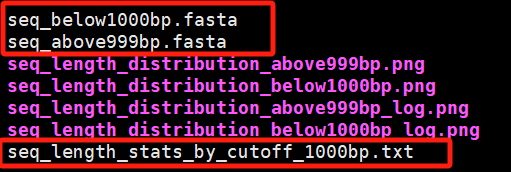
SeqLengthPlot是一种一体化序列长度分析与可视化工具,其主要功能是可对fasta文件进行统计。此外,还可对序列进行自定义长度的拆分,得到两种长度的序列文件(“长序列”和“短序列”)。同时,在分析过程中可进行直方图,直观展示数据特征。 图 软件工作流程 2 软件安装 直接Clone到分析服务器的目录之中 git clone https://github.com/danydguezperez/SeqLengthPlot.git 进入SeqLengthPlot目录 cd SeqLengthPlot/ 执行命令 python SeqLengthPlot_v2.0.2.py -i input.fasta 3 实例展示 执行软件命令 python SeqLengthPlot_v2.0.2.py -i Assembly_Ss_SE.Trinity.fasta -o output --cutoff 1000 –nt --showplot 发现直接执行上述命令会发生错误 原因是TkAgg是Matplotlib 的交互式后端,依赖 tkinter(Python的GUI 库)。在 Linux 服务器等无显示器的环境中,GUI不可用,导致此错误。这里解决的方案是使用非交互式后端(如Agg),并执行以下命令: python SeqLengthPlot_v2.0.2.py -i Assembly_Ss_SE.Trinity.fasta -o output --cutoff 1000 --nt --showplot --backend Agg 成功,无报错信息,获取结果文件。 参数信息 基本选项: ✔ -h, --help:显示帮助信息; ✔ -i INPUT, --input INPUT:必选参数,指定输入FASTA文件的路径; ✔ -o OUTPUT, --output OUTPUT:指定输出文件的目录。如果没有提供,默认使用输入文件所在的目录。 序列处理选项: ✔ --cutoff CUTOFF:设置分割序列的长度阈值(默认值为200); ✔ --nt:指定输入文件包含核苷酸序列(默认选项); ✔ --prot:指定输入文件包含蛋白质序列。 可视化选项: ✔ --showplot:启用交互式显示图表(默认不显示); ✔ --backend BACKEND:设置绘图的后端引擎(默认是 TkAgg),对于Mac用户,可以指定 MacOSX 以获得更好的兼容性。 4 结果展示 以上为自定义长度≥1000bp和<1000bp序列长度分布图(PNG 格式)。 在结果文件中,还包含长度 ≥1000bp和<1000bp拆分后的fasta文件,seq_length_stats_by_cutoff_1000bp.txt文件可快速获取数据集的统计摘要。 5 与其他工具比较 参考文献 [1] Domínguez-Pérez D, Agüero-Chapin G, Leone S, Modica MV. SeqLengthPlot v2.0: an all-in-one, easy-to-use tool for visualizing and retrieving sequence lengths from FASTA files. Bioinform Adv. 2024 Nov 20;5(1):vbae183. [2] https://github.com/danydguezperez/SeqLengthPlot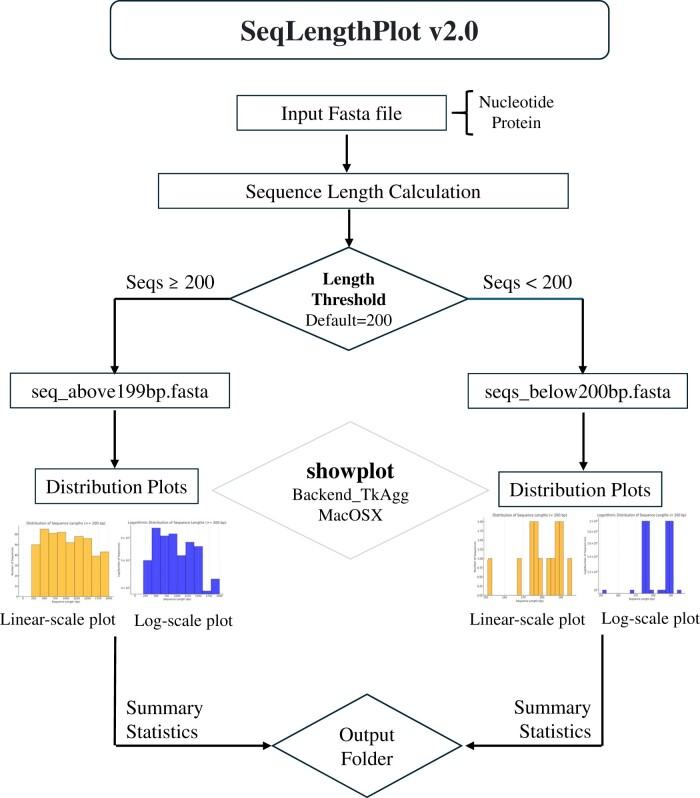






电话:021-61283010
网址:www.bio-chain.com
邮箱:marketing@bio-chain.com
地址:上海市徐汇区虹漕路421号65幢303室
上海柏辰生物科技有限公司 版权所有 严禁复制 网站备案号:沪ICP备09099502号-1 沪公网安备 31010402002899号 网站建设:铭心科技



